La rinitis alérgica (RA) es un trastorno inflamatorio crónico de la mucosa nasal mediado por respuestas de hipersensibilidad alérgica a alérgenos ambientales. Como se reportó, 10 %-20 % de la población mundial sufre de RA, la cual se convirtió en un problema de salud mundial. Sin embargo, la causa subyacente de la RA es poco conocida y la prevención de la enfermedad es imposible. La RA es una enfermedad heterogénea compleja que involucra factores genéticos y ambientales. Los estudios de gemelos demostraron que los factores genéticos desempeñan un papel importante en la patogénesis de la enfermedad alérgica y las personas con antecedentes familiares de enfermedades alérgicas son más propensas a desarrollar síntomas alérgicos. Los polimorfismos de nucleótido único (SNP) se consideran un factor genético crucial para las enfermedades alérgicas que pueden aumentar o disminuir la susceptibilidad de RA. Tang et al elucidaron que el SNP IL13 rs20541 puede contribuir a la susceptibilidad a la RA y aumentar el riesgo de RA en la población asiática. Además, Quian et al demostraron que el alelo A de rs11466651 en el TLR10 reduce la susceptibilidad de RA, lo que significa que el polimorfismo protege contra RA. Por consiguiente, la asociación de SNP con RA también es controvertida. Por ejemplo, algunos investigadores encontraron que el alelo T rs2243250 de IL-4 podría aumentar o disminuir el riesgo de RA. Por el contrario, otros creían que el alelo T no se relaciona con una mayor prevalencia de RA.
Para resolver las limitaciones de las investigaciones individuales, se requiere una evaluación integral de la relación entre los SNP y la vulnerabilidad a RA en estudios sintetizados. El propósito de este estudio fue, en primer lugar, proporcionar una revisión detallada de los SNP investigados en RA y, en segundo lugar, evaluar la correlación entre los SNP reportados con mayor frecuencia y la susceptibilidad a RA mediante metaanálisis para comprender mejor el mecanismo molecular de esta enfermedad y proporcionar objetivos o enfoques terapéuticos subyacentes para la reutilización de fármacos existentes.
2 MÉTODOS
2.1 Estrategia de búsqueda
Se realizó una búsqueda bibliográfica exhaustiva en PubMed y CNKI, con las estrategias de búsqueda (“rinitis alérgica” o “RA”) y (“mutación” o “polimorfismo”). La estrategia de búsqueda detallada se proporciona en la tabla complementaria S1. No hubo restricciones de idioma o fecha de publicación. La búsqueda bibliográfica finalizó el 11 de febrero de 2022. Dos investigadores (FX y LW) evaluaron de forma independiente la elegibilidad para esta publicación, con el propósito de revisar la literatura de la manera más amplia y eficiente posible, también revisaron referencias de estudios relevantes para complementar la literatura valiosa. En caso de duda, se adquirió el texto completo y cualquier desacuerdo se resolvió mediante el acuerdo del revisor.
2.2 Selección de estudios
Para ganar la credibilidad de los resultados y restringir la cantidad de metaanálisis completados, sólo se consideraron en el análisis los genes y polimorfismos reportados en al menos tres estudios diversos. Para el metaanálisis, los estudios cumplieron con el siguiente estándar de inclusión: 1) estudios originales de casos y controles de RA; 2) Se reportó una asociación entre RA y al menos un polimorfismo y se dispuso de datos suficientes para calcular la razón de probabilidad (OR) y el IC de 95 % correspondiente; 3) la frecuencia del genotipo no se desvió de manera significativa del equilibrio de Hardy-Weinberg (HWE); 4) de acuerdo con los síntomas y pruebas de alérgenos o confirmados de manera histológica, las personas se diagnosticaron con RA. Se excluyeron investigaciones duplicadas e investigaciones con datos superpuestos. También se excluyeron los estudios retrospectivos, las revisiones sistemáticas y los metaanálisis.
Los criterios de exclusión fueron los siguientes: 1) no ser un estudio de casos y controles; 2) irrelevante para los SNP y el riesgo de RA; 3) falta de datos detallados.
2.3 Extracción de datos
Un investigador (FX) extrajo los datos de todos los estudios elegibles, que se verificaron por el segundo investigador (LW). Las divergencias se resolvieron mediante consulta con el tercer investigador (YP). Para cada estudio se recolectaron los siguientes datos: el título, los autores, la revista, el año de publicación, el país del equipo de estudio, la capacidad de la muestra y el método de diagnóstico.
2.4 Análisis estadísticos
Después de la extracción de datos, se realizaron metaanálisis de polimorfismos en al menos tres publicaciones. Se empleó el programa RevMan 5.3 para completar todos los análisis estadísticos. Se evaluó el grado de asociación entre la RA y el polimorfismo de interés. Se aplicó un modelo de efectos fijos o de efectos aleatorios al metaanálisis completo mediante la fusión de los valores de OR (IC de 95 %) de las publicaciones involucradas. Antes de fusionar las publicaciones, se debió encontrar la heterogeneidad de manera correcta y efectiva, es decir, la prueba de heterogeneidad. La prueba I2 es el índice de evaluación de la heterogeneidad propuesto por Higgins et al. Cuando I2 = 0 indica que no se observa heterogeneidad, a mayor valor de I2 mayor heterogeneidad. La selección del modelo radica en la heterogeneidad de los interestudios. Cuando no hubo evidencia de heterogeneidad, se seleccionó el modelo de efectos fijos (I2 <50 %, p > 0.05) Si no, se utilizó un modelo de efectos aleatorios. Un valor de OR superior a 1 con un límite inferior del IC superior a 1 y p < 0.05 se cree que es significativo de forma estadística, o menos de 1 con el límite superior del IC inferior a 1.
El sesgo de publicación se juzgó de forma intuitiva al dibujar un gráfico en embudo. El gráfico en embudo simétrico, no se encontró sesgo de publicación; de lo contrario, existía sesgo de publicación. Además, se estimó de manera cuantitativa el sesgo de publicación mediante el Test de Egger. Para cada polimorfismo involucrado en esta investigación, se realizó una búsqueda de artículos para identificar metaanálisis previos, los resultados se compararon con los resultados anteriores para identificar registros potenciales.
3 RESULTADOS
3.1 Métodos de búsqueda y filtrado de datos
Después de la proyección (Figura 1), se identificaron un total de 23 polimorfismos que se reportaron en al menos 3 literaturas. Ciento cuarenta y dos artículos (Tabla complementaria S1) reportaron 78 genes diversos, y 12 de ellos se estudiaron en al menos tres publicaciones. Cincuenta y cinco polimorfismos se identificaron en total en el presente estudio (Tabla complementaria S2). Luego, se completó una revisión sistemática para cada uno de los 23 polimorfismos de cribado, los cuales se estudiaron en más de tres publicaciones (Tabla 1). El número de publicaciones además de cada polimorfismo, el grado de heterogeneidad, la cantidad de pacientes y controles, el OR [IC 95%] y la aplicación de un modelo de efectos aleatorios o de efectos fijos se muestran en la Tabla 1.
3.2 Resultados del metaanálisis
TNF_rs1800629 (-308G/A)
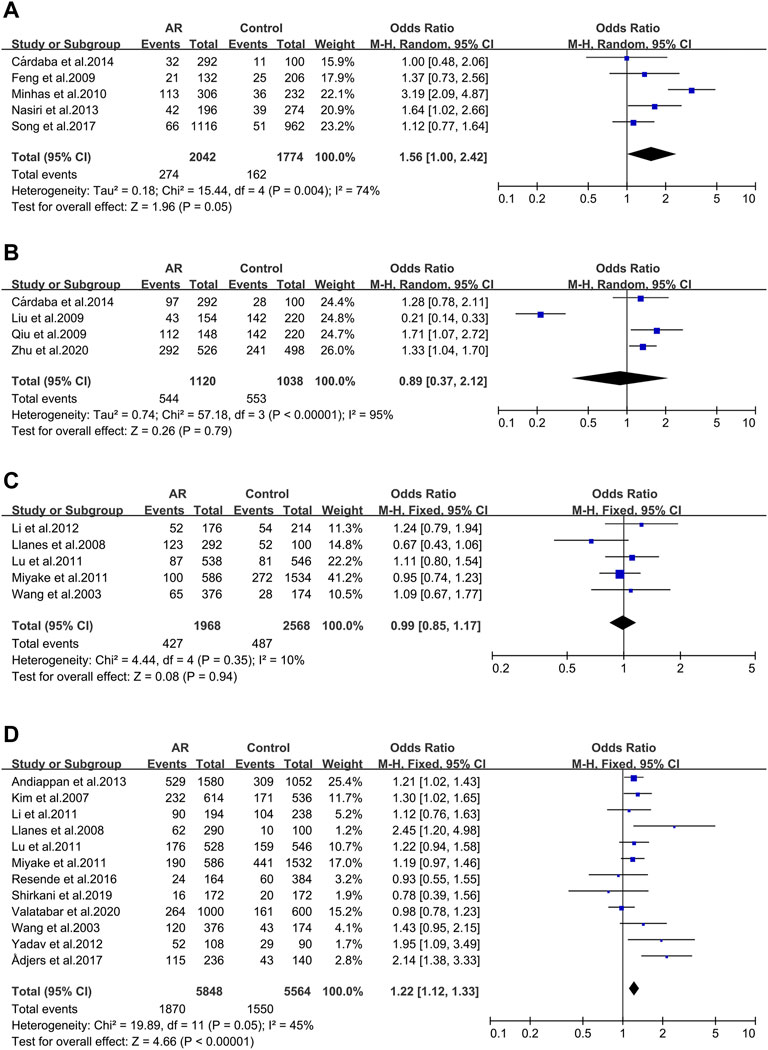
Cinco estudios evaluaron seis variantes del factor de necrosis tumoral (TNF), de las cuales sólo se consideró el TNF RS1800629. Se incluyeron cinco publicaciones en este metaanálisis. No se observaron asociaciones significativas (OR 1.56; IC 95% 1.00-2.42) al aplicar un modelo de efectos aleatorios (Figura 2A), y el sesgo de publicación tampoco se observó (t = -0.4; p = 0.717) (Figura complementaria S1A).
TGFβ1 _ rs1800469 (-509C/T)
Se incluyeron cuatro estudios del polimorfismo rs1800469 del factor de crecimiento transformante β1 (TGFβ1) (32/65/104/141). No se observaron asociaciones significativas (OR 0.89; IC 95% 0.37-2.12) al aplicar un modelo de efectos fijos (Figura 2B) y el sesgo de publicación tampoco se observó (t = -0.51; p = 0.659) (Figura complementaria S1B).
3.2.1 El gen IL-13
Se seleccionaron trece artículos de texto completo sobre SNP de interleucina 13 (IL-13) de interés. Se analizaron dos variantes genéticas de IL-13: rs1800925 (Figura 2C), rs20541 (Figura 2D).
IL-13 _ rs1800925 (-1112C > T/1055). IL-13 _ rs1800925 (-1112C > T/1055) se consideró en cinco estudios y se completó un metaanálisis sobre ellos. No se observaron asociaciones significativas (OR 0.99; IC de 95 % 0.85-1.17) en un modelo de efectos fijos (Figura 2C), y el sesgo de publicación tampoco se observó (t = 0.01; p = 0.994) (Figura complementaria S1C).
IL-13_rs20541 (130/+ 2044G > A). Para el metaanálisis se consideraron doce estudios relacionados con el alelo IL-13_rs20541 A. Se observó asociación (OR 1.22; IC de 95% 1.12-1.33) al aplicar un modelo de efectos fijos (Figura 2D) sin sesgo de publicación (t = 1.19; p = 0.262) (Figura complementaria S1D).
3.2.2 El gen CTLA-4
Después de la selección, se analizaron cuatro de los nueve mutantes alélicos principales del antígeno 4 asociado a linfocitos T citotóxicos (CTLA-4): rs3087243 (Figura 3A), rs231725 (Figura 3B), rs11571302 (Figura 3C) y rs11571315 (Figura 3D) y sólo rs11571302 se asoció de manera significativa con RA.
CTLA-4 _ rs3087243. Se incluyeron tres estudios. No se observaron asociaciones significativas (OR 1.06; IC de 95%: 0.93-1.20) al aplicar un modelo de efectos fijos (Figura 3A). Y tampoco se observó asimetría en la prueba de gráfico en embudo para sesgo (t = -3.7; p = 0.168) (Figura complementaria S1E).
CTLA-4 _ rs231725. Tres estudios participaron en el metaanálisis. No se encontraron asociaciones significativas (OR 0.81; IC de 95% 0.63-1.05) al aplicar un modelo de efectos aleatorios (Figura 3B) y el sesgo de publicación tampoco se observó (t = - 4.81; p = 0.13) (Figura complementaria S2F).
CTLA-4 _ rs11571302. La variante CTLA-4 (rs11571302) se consideró en tres estudios. Cuando se consideró el alelo A como el alelo de riesgo, se encontró una correlación (OR 1.41; IC 95% 1.24-1.60) en un modelo de efectos fijos (Figura 3C) sin sesgo de publicación (t = -10.67; p =0.059) (Figura complementaria S2G).
CTLA-4_rs11571315
Se consideraron tres estudios. No se observaron asociaciones significativas (OR 1.00; IC de 95 %: 0.89-1.55) en un modelo de efectos fijos (Figura 3D) y el sesgo de publicación tampoco se vio (t = 4.29; p = 0.146) (Figura complementaria S2H).
3.2.3 El gen IL-4
En los 8 estudios de RA y polimorfismos de interleucina-4 (IL-4) se reportaron cinco variantes de IL-4. Sólo tres de ellos se consideraron.
IL-4 _ rs2243250 (−590C > T/589). IL-4 _ rs2243250 se consideró en ocho estudios. Cuando se consideró el alelo T como el alelo de riesgo, no se identificaron asociaciones (OR 1.37; IC de 95 %: 0.95-1.98) en un modelo de efectos aleatorios (Figura 4A) y el sesgo de publicación tampoco se vio (t = 0.71; p = 0.506) (Figura complementaria S2A).
IL- 4 _ rs2227284 (T2979G). También se consideraron tres estudios y se completó un metaanálisis de estos estudios. No se encontraron asociaciones significativas (OR 1.56; IC de 95 % 0.64-3.80) en un modelo de efectos aleatorios (Figura 4B) y el sesgo de publicación tampoco se observó (t = 1.64; p = 0.349) (Figura complementaria S2B).
IL-4 _ rs2070874(C-33T). En este metaanálisis se incluyeron tres estudios. No se observaron asociaciones significativas (OR 0.68; IC de 95 % 0.34-1.37) al aplicar un modelo de efectos aleatorios (Figura 4C) y el sesgo de publicación tampoco se detectó (t = -2.24; p = 0.267) (Figura complementaria S2C).
Al considerar los 10 estudios de variantes de CD14 y RA, sólo el polimorfismo rs2569190 se ajustaba al estándar de inclusión. Se agruparon las diez publicaciones y no se encontraron asociaciones significativas (OR 0.93 IC de 95% 0.78-1.12) al aplicar un modelo de efecto (Figura 4D), así como el sesgo de publicación (t =1.71; p = 0.126) (Figura complementaria S2D).
3.2.4 El gen FOXP3
Se analizaron dos de las cuatro variantes genéticas principales de la caja de la cabeza del tenedor P3 (FOXP3): rs3761548 (Figura 5A) y rs2232365 (Figura 5 B) y ninguno se asoció de forma significativa con RA.
FOXP3_ rs3761548 (−3279A > C). Se consideraron cinco estudios para el metaanálisis. No se identificó asociación (OR 0.89; IC de 95 % 0.65-1.20) al emplear el modelo de efectos aleatorios (Figura 5A) y no se encontró sesgo de publicación (t = 0.12; p = 0.910) (Figura complementaria S2E).
FOXP3_ rs2232365 (−924A > C). Cuatro estudios se involucraron (Figura complementaria S26A). No se encontraron asociaciones significativas (OR 0.97; IC de 95 % 0.83-1.13) en un modelo de efectos fijos (Figura 5B) y el sesgo de publicación tampoco se encontró (t = 0.82; p = 0.498) (Figura complementaria S2F).
3.2.5 El gen IL-18
Se reportaron siete variantes de interleucina-18 (IL-18). Sólo tres de ellos se consideraron.
IL-18_ rs1946518 (−607C/A). El metaanálisis se completó con datos de cuatro publicaciones. No se encontraron asociaciones significativas (OR 1.02; IC de 95% 0.90-1.16) al aplicar un modelo de efectos fijos (Figura 5C) y el sesgo de publicación no se detectó en el gráfico en embudo (t = 2.96; p = 0.098) (Figura complementaria S3A).
IL-18 _ rs187238 (−137G/C). En este metaanálisis se consideraron tres estudios relevantes. No se observaron asociaciones significativas (OR 1.10; IC de 95 % 0.94-1.28) en un modelo de efectos fijos (Figura 5D) y no se observó sesgo de publicación (t = 2.96; p = 0.098) (Figura complementaria S3B).
IL-18_rs360721(C133/140G). Tres estudios participaron en el metaanálisis. No se observaron asociaciones significativas (OR 0.89; IC de 95 % 0.77-1.04) al aplicar el modelo de efectos fijos (Figura 5E). No se identificó ningún posible sesgo de publicación (t =10.68; p = 0.059) (Figura complementaria S3C).
3.2.6 El gen Tim-3
Después de la selección, sólo se analizó uno de los principales mutantes alélicos del dominio de inmunoglobulina de las células T y la proteína 3 del dominio de mucina (Tim-3).
Tim-3_ rs10515746 (−574G/T). Cuatro estudios participaron en este metaanálisis. No se encontraron asociaciones significativas (OR 1.21; IC de 95 % 0.78-1.88) en un modelo de efectos fijos (Figura 6A) y el sesgo de publicación tampoco se identificó (t =2.84; p =0.105) (Figura complementaria S3D.
3.2.7 El gen IL-4R
Después de la selección de datos, se analizaron los dos mutantes alélicos primarios de IL-4R.
IL-4R_ rs1801275 (Q576R/Gln551Arg). En este metaanálisis se consideraron nueve estudios. Se observaron asociaciones significativas (OR 1.48; IC de 95 % 1.07-2.05) al aplicar un modelo de efectos aleatorios (Figura 6B) y se detectó un sesgo de publicación entre los estudios (t = 2.62; p = 0.034) (Figura complementaria S4A).
IL-4R_ rs1805010 (Ile50Val). Se incluyeron tres estudios para esta variante. No se identificó ninguna asociación (OR 0.84; IC de 95% 0.63-1.13) (Figura 6C) sin sesgo de publicación (t =0,51; p =0.699) (Figura complementaria S4B).
3.2.8 El gen ACE
ACE_ I/D. Cuando se agruparon los cinco estudios, se observó una asociación significativa en un modelo de efectos aleatorios (OR 1.47; IC de 95 % 1.09–1.97) (Figura 6D). Y no se identificó sesgo de publicación (t = 1.25; p = 0.301) (Figura complementaria S4C).
3.2.9 El gen TAP1
TAP1_333V/I. Se identificaron cuatro estudios relacionados con ACE y RA. Según estos cuatro estudios restantes, no se encontraron asociaciones significativas (OR 1.26; IC de 95 %: 0.50-3.15) en un modelo de efectos aleatorios (Figura 7A) y tampoco se detectó ningún posible sesgo de publicación (t = -0.57; p = 0.627) (Figura complementaria S4D).
TAP1_637G/D. De los cuatro estudios seleccionados para TAP1_637G/D, el metaanálisis se completó con información de estas publicaciones y no se encontraron asociaciones significativas (OR 0.72; IC de 95 % 0.31-1.69) al emplear un modelo de efectos aleatorios (Figura 7B), así como el sesgo de publicación (t = 0.00; p = 0.999) (Figura complementaria S4E).
4 DISCUSIÓN
El objetivo de esta investigación fue ofrecer una revisión detallada de los polimorfismos sobre RA y realizar un metaanálisis de los polimorfismos más estudiados. Al final, se analizaron 23 polimorfismos (en 12 genes). Se identificaron cuatro polimorfismos que se relacionaron de forma significativa con el riesgo de RA. Por lo tanto, se permitió identificar aquellos polimorfismos que tenían el potencial de constituir una prueba de detección para RA y proponer combinaciones de SNP que podrían reconocer RA de forma previa: IL-13 rs20541, CTLA-4 rs11571302, IL-4R rs1801275 y ACE (I/ D).
Para ser lo más detallados posible, se realizaron consultas extensas de búsqueda en PubMed y CNKI. Se encontró que la cantidad de 142 artículos (que involucraban 78 genes) se relacionaban. Para aumentar la eficiencia, en el estudio sólo se consideraron los genes que se investigaron en tres o más estudios. De las 142 publicaciones citadas en la sección de resultados, 38 se aumentaron de forma manual debido a que faltaba la palabra clave “RA”. El aumento manual de referencias no fue inesperado, ya que la estrategia permitió encontrar datos relevantes de manera rápida y precisa. Hasta donde se sabe, este estudio es el primero en enfatizar la posible asociación entre RA y los polimorfismos de CTLA- 4 rs11571302 por medio de metaanálisis (Figura 2C). El gen CTLA-4 desempeña un papel regulador negativo en la activación de las células T presentadoras de antígenos (CPA). La infraexpresión inducible de CTLA-4 con desequilibrio Th2/Treg se asocia a RA. En este metaanálisis, CTLA-4 rs11571302 se relacionó a RA (con OR de 1.41 [IC de 95% 1.24-1.60]). Las frecuencias del alelo rs11571302 A se asociaron con el riesgo de RA. En contraste con el resultado de CTLA-4 rs11571302, los datos de la literatura sobre CTLA-4 rs3087243, CTLA-4 rs231725 y CTLA-4 rs11571315 muestran que no tienen una asociación notable con el riesgo de RA.
Se sabe que la inflamación y la inmunización participan en la patogenia de la RA. Las citocinas tipo 2 (IL-13, IL-4 e IL-18) son los factores iniciadores de la respuesta inflamatoria de las vías respiratorias. Varios estudios revelaron que los niveles de IL-13, IL4 e IL18 en la secreción periférica parecen estar elevados de manera anormal en pacientes con RA. El metaanálisis enfatizó una asociación entre el polimorfismo rs20541 de IL-13 y el riesgo de RA. IL-13 rs20541 imparte susceptibilidad al desarrollo de RA. Sin embargo, no se observó ningún vínculo entre IL-13 rs1800925 y RA. Estos resultados fueron similares a los metaanálisis anteriores.
No se detectaron asociaciones con RA para IL-4rs2243250, IL-4 rs2227284 e IL-4 rs2070874. Por el contrario, un estudio previo reportó que el genotipo IL-4 rs2243250 se asocia con la susceptibilidad a RA. Se excluyó un estudio que ya no se publicó en línea y que se tuvo en cuenta en el metaanálisis anterior. De manera obvia, la selección de participantes de control afectó el resultado final. Al considerar la inestabilidad de los resultados, se requieren más estudios para obtener un resultado más completo para asegurar o refutar la asociación subyacente entre RA y el polimorfismo rs2243250 de IL-4. Tampoco se encontraron asociaciones para IL-18 rs1946518, IL-18 rs187238 e IL-18 rs360721, lo que se confirmó con el metaanálisis anterior. Investigaciones adicionales se consideran innecesarias.
Todas las funciones biológicas de la IL-4 están mediadas por el efector IL-4R, incluida la mediación del desequilibrio Th1/Th2, la promoción de la proliferación de células B y la síntesis de IgE, que desempeñan un papel crucial en la rinitis alérgica. IL-4R rs1801275 se asoció con el riesgo de RA (OR 1.48; IC de 95 % 1.07-2.05) y el alelo R aumenta la susceptibilidad a la RA. Pero la correlación debe validarse en más estudios, ya que existe un posible sesgo de publicación en la prueba de Egger. Por el contrario, no se detectó ninguna asociación entre RA y el polimorfismo rs1805010 de IL-4R.
El polimorfismo ACE I/D puede influir en la fuerza de la respuesta inmunológica y se reportó que se asocia con trastornos atópicos, enfermedades inflamatorias. Los resultados revelaron que el alelo ACE D aumenta el riesgo de desarrollar RA, de acuerdo con investigaciones previas.
El TNF-α funciona como una citocina proinflamatoria que participa en la activación de las células T y la liberación de citocinas, el reclutamiento de neutrófilos, macrófagos y monocitos, y el aumento de la liberación de histamina de las células cebadas de las vías respiratorias. Se aseguró el resultado conocido de asociación mostrado de forma previa por otro metaanálisis. No se encontró correlación entre RA y TNF-α (−308G/A).
Se sabe que el TGF-β participa en la RA, que puede inducir a las células T CD4+ iniciales a diferenciarse en células Treg CD4+ CD25+ y estabilizar la función inmunitaria del cuerpo. Los niveles elevados de TGF-β1 pueden aliviar la inflamación de la mucosa nasal en la RA. Los resultados que se centraron en la asociación entre el polimorfismo TGF-β1 rs1800469 y el riesgo de RA fueron inconsistentes. Un artículo demostró que el polimorfismo TGF-β1 tiene un efecto protector para la RA. Por el contrario, los resultados de otras investigaciones mostraron que el SNP TGF-β1 podría aumentar la susceptibilidad de desarrollar RA o no se asociaba con RA. En este trabajo no se encontró una relación entre RA y el polimorfismo TGF-β1 rs1800469. Se necesitan investigaciones adicionales de casos y controles bien diseñadas en grandes cohortes para confirmar el vínculo subyacente entre RA y el polimorfismo TGF-β1 rs1800469.
Los estudios demostraron que CD14/-159 puede influir en la regulación de la expresión del gen CD14 y por lo tanto, modular la influencia de los eventos mediados por CD14 en las respuestas inmunitarias innatas y adaptativas. CD14 se une forma específica a LPS para apoyar la conversión de Th0 a Th1 y las citocinas producidas por Th1 y Th2 controlan la síntesis de IgE. En comparación con las personas normales, el CD14 sérico en pacientes con RA es mayor de manera significativa y el riesgo de RA se asocia con los portadores homocigotos del alelo T (genotipo TT). Se reportó que los portadores del genotipo TT tienen un aumento significativo en los niveles séricos de sCD14 y una disminución concomitante en la IgE sérica total. Sin embargo, la expresión de homocigotos TT fue mayor en pacientes con RA, pero no se asoció con la expresión de IgE entre la población china. En este metaanálisis, el alelo T de CD14 (−159C/T) no se asoció con la susceptibilidad a la RA, lo que concordaba con estudios previos.
FOXP3, como factor de transcripción específico de las células Treg, se involucra en el desarrollo y el mantenimiento funcional de las células Treg. Comparado con las personas sanas, la expresión de FOXP3 en la sangre periférica de los pacientes con RA disminuye de manera significativa, lo que conduce a la disfunción de las células Treg y es un factor importante que conduce a la aparición de la RA. El resultado fue similar al metaanálisis anterior. No hubo vínculo entre RA y FOXP3 rs3761548 o FOXP3 rs2232365.
Tim-3 tiene un efecto regulador negativo sobre las células Th1 y provoca un desequilibrio Th1/Th2, que es necesario para el desarrollo de RA. Se reportaron niveles incrementados de Tim-3 en la sangre periférica de pacientes con RA. Sin embargo, el resultado del metaanálisis de Tim-3 rs10515746 indicó que no se relacionó de manera significativa con el riesgo de RA.
El antígeno inhalado se presenta a las células T CD4 + en la RA, y el gen TAP1 codifica las moléculas involucradas en el procesamiento de antígenos endógenos. El polimorfismo del gen TAP puede afectar los procesos de selección y transporte de péptidos antigénicos y cambiar la regulación de la respuesta inmune en la RA. Sin embargo, no se identificó el vínculo potencial entre RA y TAP1 333V/I o TAP1 637G/D.
Al final, este estudio tiene varias limitaciones. En primer lugar, para una mayor eficiencia, sólo consideró los polimorfismos citados en más de tres estudios diferentes, lo que puede pasar por alto algunas asociaciones significativas con RA. Por lo tanto, para confirmar las correlaciones subyacentes entre los polimorfismos restantes y la susceptibilidad de RA, serán necesarios más estudios. En segundo lugar, la susceptibilidad a la RA no sólo se relaciona con la herencia, sino también con la raza, la edad, el sexo, etc. Sin embargo, debido al número pequeño de estudios y muestras pequeñas en el presente análisis, no se realizaron estratificaciones por raza, edad y sexo. Además, la susceptibilidad a RA puede afectarse por múltiples locus, que se coordinan entre sí para compensar la susceptibilidad genética para desarrollar RA. Estas pueden ser las razones de porqué sólo se observaron 4 SNP que tienen asociación con el riesgo de RA en este estudio.
5 CONCLUSIÓN
Como se mencionó antes, cuatro de los polimorfismos podrían analizarse de manera simultánea ya que se asociaron con RA. Para los pocos polimorfismos no involucrados en este estudio, es decir, aquellos que mostraron una correlación positiva basada en dos estudios, es necesaria investigación adicional. Este trabajo permite a los investigadores comenzar a encontrar los marcadores subyacentes para desarrollar un método de diagnóstico para pacientes con RA. El panel de estos polimorfismos exhibe el potencial de formar una prueba de detección genética sobre RA y, por lo tanto, puede reducir el tiempo utilizado en el diagnóstico de RA.
Xiang F, Zeng Z, Wang L, Yang YP, Zhang QX. Polymorphisms and AR: A Systematic Review and Meta-Analyses. Front Genet. 2022 Jul 1;13:899923. doi: 10.3389/fgene.2022.899923.
Centro Regional de Alergia e Inmunología Clínica CRAIC, Hospital Universitario “Dr. José Eleuterio González” UANL, Monterrey, México
Dra. Med. Sandra Nora González Díaz Jefe y Profesor
Dra. Med. Rosa Ivett Guzmán Avilán Profesor
Dra. Claudia Anai Estavillo Gómez Residente 1er Año
Dra. Alejandra Macías Weinmann Profesor




No hay comentarios:
Publicar un comentario
Nota: solo los miembros de este blog pueden publicar comentarios.